Subiecte
Abstract
Stocarea de date bazată pe ADN a apărut ca o metodă promițătoare pentru a satisface cererea în creștere exponențială de stocare a informațiilor. Cu toate acestea, implementarea practică a stocării datelor pe bază de ADN rămâne o provocare din cauza costului ridicat al scrierii datelor prin sinteza ADN-ului. Aici, propunem utilizarea bazelor degenerate ca caractere de codificare în plus față de A, C, G și T, care mărește cantitatea de date care poate fi stocată pe lungimea secvenței de ADN proiectate (capacitatea informațională) și scăderea cantității de ADN sinteză pe stocarea datelor unitare. Folosind metoda propusă, am obținut experimental o capacitate de informație de 3,37 biți/caracter. Capacitatea de informare demonstrată este de peste două ori în comparație cu cea mai mare capacitate de informare atinsă anterior. Metoda propusă poate fi integrată cu tehnologiile sintetice în viitor pentru a reduce costul stocării datelor pe bază de ADN cu 50%.
Introducere
Aici, propunem și demonstrăm utilizarea bazelor degenerate (combinație a celor patru baze ADN care pot fi inserate la orice site-uri de bază dintr-o secvență) 11 ca caractere de codificare suplimentare pentru a depăși limita de capacitate de informații teoretice de 2,0 biți/caracter. Bazele degenerate sunt localizate în secvența ADN atunci când nucleotidele sunt amestecate la o poziție specifică în secvența ADN. De exemplu, în secvența „AWC”, „W” indică o combinație de A și T; astfel, există două tipuri de variante de nucleotide în grupul de molecule: „AAC” și „ATC”. În acest articol, utilizând unsprezece baze degenerate în plus față de cele patru caractere ADN, realizăm experimental o capacitate de informație de 3,37 biți/caracter în biblioteca oligonucleotidelor care cuprinde sute de copii ale fiecărei secvențe. Cu alte cuvinte, stocăm mai multe date folosind mai puține copii ale fiecărei secvențe, comparativ cu numărul de molecule utilizate în studiile anterioare. Ca rezultat, demonstrăm că lungimea ADN necesară pentru a stoca aceeași cantitate de date a fost redusă cu mai mult de jumătate comparativ cu rapoartele anterioare 3,4,5,6,9,10. Tehnologia propusă poate fi integrată cu tehnologiile sintetice în viitor pentru a reduce costul stocării datelor pe bază de ADN cu 50%.
Rezultate
Adăugarea de baze degenerate la stocarea datelor pe bază de ADN
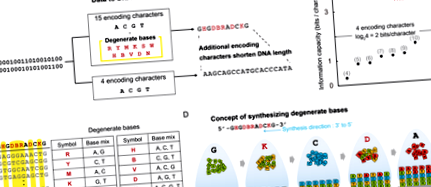
Stocarea de date bazată pe ADN, cu adăugarea de baze degenerate, permite o creștere a capacității informaționale. (A) Datele binare sunt codificate în secvențe de ADN care cuprind nu numai cele 4 caractere tradiționale de codificare A, C, G și T, ci și 11 baze degenerate suplimentare. Lungimea ADN-ului codificat este mai mică decât cea a metodei de codificare cu patru caractere. (B) Limita de capacitate a informațiilor teoretice este, prin urmare, mărită de la 2 biți/caracter la 3,9 biți/caracter. Punctele din grafic descriu valorile capacității informaționale din cercetările anterioare, iar numerele indică referința corespunzătoare. (C) O bază degenerată reprezentată de un caracter de codificare descrie un bazin mixt de mai mult de două tipuri de nucleotide. (D) Bazele degenerate pot fi generate prin amestecarea fosforamiditelor ADN în timpul sintezei.
Structura și rezultatul decodării platformei de stocare a datelor pe bază de ADN
Structura și rezultatul decodării platformei de stocare a datelor pe bază de ADN. Am obținut cea mai mare capacitate informațională și densitatea fizică a stocării datelor bazate pe ADN. (A) Structura de proiectare a fragmentelor de ADN. (B) Fragmentele de ADN pot fi analizate folosind NGS. După clasificare după adresă, bazele degenerate pot fi decodificate examinând distribuția caracterelor în aceeași poziție (bara galbenă). (C) Bazele degenerate pot fi determinate din graficul de dispersie al raportului bazelor în aceeași poziție. (D) Rata de eroare a bazelor de ADN determinate în acoperirea medie specifică a fragmentelor totale. Abaterile standard (s.d.) au fost obținute prin repetarea eșantionării aleatorii de 10 ori. Barele de eroare reprezintă s.d. (E) Rezumatul rezultatelor experimentale. Capacitatea informațională este calculată din informațiile de intrare în biți împărțite la numărul de caractere de codificare (exclusiv cea a site-urilor adaptorului). Am comparat rezultatele muncii noastre cu cele ale lui Erlich și Zielinski 10, care au raportat anterior cea mai mare capacitate informațională și densitate fizică folosind sinteza oligo cumulată și date de secvențializare de mare viteză. Densitatea fizică este raportul dintre numărul de octeți codificați și greutatea bibliotecii ADN utilizate pentru a decoda informațiile.
Verificarea și proiecția costurilor platformei propuse prin simulare
0,05 $/100 nt, Notă suplimentară) 22 utilizând sintetizator bazin oligonucleotidic pe bază de jet de cerneală. Mai mult, din moment ce costul secvențierii ADN-ului scade mai repede decât legea lui Moore și mai rapid decât cel al sintezei ADN-ului, diferența de preț dintre secvențierea și sinteza va crește în ordine, dacă tendința actuală continuă cu 1,23. Când se aplică acest cost, chiar dacă platforma propusă are 2000x acoperire NGS ca caz extrem, costul citirii datelor va fi mai mic de 5% din costul de scriere și mai mic de 0,5%, ceea ce va fi neglijabil, în cinci ani (Fig. 3B). Presupunând că sintetizatorul oligonucleotidic pe bază de jet de cerneală este setat pentru sinteza degenerată a bazei, sa estimat că platforma propusă reduce costul stocării datelor pe bază de ADN la 2052 USD/1 MB când se utilizează 15 caractere de codificare și 1795 USD/1 MB când se utilizează 21 de caractere de codificare, care reprezintă aproximativ 50% din minimul anterior de 3555 USD/1 MB 10 (Fig. 3B, Notă suplimentară).
Discuţie
În această demonstrație, utilizând baze degenerate, capacitatea informațională și densitatea fizică au fost mai mult decât dublate comparativ cu cele ale platformelor de stocare a datelor raportate anterior pe bază de ADN. În special, pe măsură ce crește capacitatea informațională, platforma scurtează lungimea ADN-ului necesar pentru a stoca o cantitate echivalentă de date și reduce costul total al stocării datelor la jumătate. Densitatea fizică va fi crescută empiric în cercetările viitoare și vor fi urmate studii care împing limita superioară a densității fizice. De asemenea, metoda introdusă reduce timpul de sinteză, dacă este disponibil un sistem de sinteză adecvat. De exemplu, tehnica de sintetizare a oligonucleotidelor pe coloană care folosește etapele de spălare, deprotejare, care crește proporțional cu lungimea oligonucleotidelor care urmează a fi sintetizate. Deoarece putem scurta lungimea sintezei pentru stocarea aceleiași cantități de date, timpul de sinteză va fi redus.
Material si metode
Codificarea datelor în secvența ADN
Pentru prima demonstrație, a fost codificat ADN un fișier text (txt) care descrie o scurtă introducere și o listă de membri ai laboratorului căruia îi aparține autorul corespunzător (Fig. S1). Pentru a doua demonstrație, a fost codificată o imagine în miniatură a manuscrisului Hunminjeongum (Fig. S2). Fișierul imagine a fost redimensionat la 692 × 574 și dimensiunea fișierului a fost de 135.393 octeți. Datele binare au fost extrase din fișier și grupate ca lungime a fragmentului de ADN. Au fost adăugate fragmente de redundanță Reed-Solomon pentru a doua demonstrație. După aceea, adresa a fost atașată. Toate cifrele au fost transformate în codoni ADN așa cum este descris în tabelele S1 - S3. Mai multe detalii ale datelor pentru codificarea ADN sunt descrise în nota suplimentară.
Pregătirea și cuantificarea probelor de ADN
Amplificarea și secvențierea ADN-ului
Probele au fost amplificate folosind qPCR (FAST 7500, Applied Biosystems) și KAPA HiFi Library Amplification Kit. Amestec de probă de 10 amestecuri masterL master, 6 uL de apă de calitate PCR, 1 uL dintr-un stoc de primer de 10 uM de Forward și Reverse fiecare, 1 uL soluție de oligo pool, 20X SYBR Green a fost utilizat. Am urmat protocolul termic standard din manual. Am verificat graficul de amplificare folosind qPCR. De îndată ce complotul a atins saturația, am oprit mașina și am purificat eșantionarea folosind kitul de purificare PCR (Qiagen). Am secvențiat piscina oligo amplificată folosind un Miniseq folosind un protocol de citire de 300 de cicluri.
ADN la decodarea datelor
Citirile de perechi ale fișierului NGS brut (format Fastq) au fost cusute folosind PEAR. După aceea, citirile NGS cu lungimile corespunzătoare au fost filtrate și citirile duplicate au fost eliminate. Citirile duplicate au fost eliminate și reprezentarea secvenței (include baza degenerată) a fost calculată. Din secvența reprezentativă, codonul ADN a fost transformat în cifre, urmând tabelele suplimentare S1 - S3. Corectarea erorilor folosind codul Reed-Solomon a fost efectuată pentru a doua demonstrație. Mai multe detalii despre decodarea ADN către date sunt descrise în nota suplimentară.
Simulare Monte Carlo
Disponibilitatea datelor
Seturile de date utilizate și/sau analizate în timpul studiului actual sunt disponibile de la autorul corespunzător, la o cerere rezonabilă.
Referințe
Zhirnov, V., Zadegan, R. M., Sandhu, G. S., Church, G. M. & Hughes, W. L. Memoria acidului nucleic. Nat. Mater. 15, 366-370 (2016).
Clelland, C. T., Risca, V. & Bancroft, C. Ascunderea mesajelor în microdoturile ADN. Natură 399, 533–534 (1999).
Bancroft, C., Bowler, T., Bloom, B. & Clelland, C. T. Depozitarea pe termen lung a informațiilor în ADN. Știință (80-.). 293, 1763c - 1765 (2001).
Goldman, N. și colab. Către stocarea informațiilor practice, de mare capacitate, cu întreținere redusă în ADN sintetizat. Natură 494, 77-80 (2013).
Church, G. M., Gao, Y. & Kosuri, S. Următoarea generație de stocare a informațiilor digitale în ADN. Ştiinţă 337, 1628 (2012).
Bornholt, J. și colab. Un sistem de stocare arhivistică bazat pe ADN - Microsoft. Cercetare. Revizuirea sistemelor de operare ACM SIGOPS 50, 637–649 (2016).
Blawat, M. și colab. Corecție de eroare înainte pentru stocarea datelor ADN. Procedura de calcul. Știință. 80, 1011–1022 (2016).
Organick, L. și colab. Acces aleatoriu la stocarea de date ADN la scară largă. Nat. Biotehnologie, https://doi.org/10.1038/nbt.4079 (2018).
Grass, R. N., Heckel, R., Puddu, M., Paunescu, D. & Stark, W. J. Conservare chimică robustă a informațiilor digitale despre ADN în silice cu coduri de corectare a erorilor. Angew. Chem. Int. Ed. Engleză. 54, 2552–5 (2015).
Erlich, Y. & Zielinsk, D. Fântâna ADN permite o arhitectură de stocare robustă și eficientă. Ştiinţă (80-.), 950-954 (2017).
Cornish-Bowden, A. Nomenclatura pentru baze specificate incomplet în secvențe de acid nucleic: recomandări 1984. Acizi nucleici Res. 13, 3021–30 (1985).
Beaucage, S. L. și Iyer, R. P. Progrese în sinteza oligonucleotidelor prin abordarea fosforamidită. Tetraedru 48, 2223–2311 (1992).
LeProust, E. M. și colab. Sinteza bibliotecilor de înaltă calitate a oligonucleotidelor lungi (150mer) printr-un nou proces de depurinare controlat. Acizi nucleici Res. 38, 2522–2540 (2010).
Cleary, M. A. și colab. Producerea de biblioteci complexe de acid nucleic folosind foarte paralel in situ sinteza oligonucleotidelor. Nat. Metode 1, 241–248 (2004).
Hughes, T. R. și colab. Profilarea expresiei utilizând microarrays fabricate de un sintetizator oligonucleotidic cu jet de cerneală. Nat. Biotehnologie. 19, 342–347 (2001).
BioSisteme aplicate. Evaluarea și izolarea oligonucleotidelor sintetice - Ghidul complet. (1992).
Hecker, K. H. și Rill, R. L. Analiza erorilor polinucleotidelor sintetizate chimic. Biotehnică 24, 256-60 (1998).
Airaksinen, A. & Hovi, T. Compozițiile de bază modificate în poziții degenerate ale unei oligonucleotide mutagene sporesc randomitatea în mutageneza de saturație a sitului. Acizi nucleici Res. 26, 576-581 (1998).
Kosuri, S. & Church, G. M. Sinteza ADN de novo la scară largă: tehnologii și aplicații. Nat. Metode 11, 499–507 (2014).
Aird, D. și colab. Analiza și minimizarea prejudecății amplificării PCR în bibliotecile de secvențare Illumina. Genomul Biol. 12, R18 (2011).
Williams, R. și colab. Amplificarea bibliotecilor genetice complexe prin emulsie PCR. Nat. Metode 3, 545–550 (2006).
Wetterstrand, K. Costuri de secvențiere a ADN-ului: date din programul de secvențiere a genomului NHGRI (GSP). Natl. Zumzet. Genom Res. Inst.
Carr, P. A. & Church, G. M. Ingineria genomului. Nat. Biotehnologie. 27, 1151–1162 (2009).
Zhang, Y. și colab. Un organism semi-sintetic care stochează și recuperează informații genetice crescute. Natură 551, 644–647 (2017).
Mulțumiri
Această lucrare a fost susținută de Centrul de finanțare Samsung Research al Samsung Electronics sub numărul proiectului SRFC-IT1601-08.
Informatia autorului
Afilieri
Departamentul de Inginerie Electrică și Calculatoare, Universitatea Națională Seoul, 1, Gwanak-ro, Gwanak-gu, Seul, 08826, Republica Coreea
Yeongjae Choi, Taehoon Ryu, Hansol Choi, Hansaem Lee, Jaejun Park și Sunghoon Kwon
Program interdisciplinar pentru bioinginerie, Universitatea Națională din Seul, 1, Gwanak-ro, Gwanak-gu, Seul, 08826, Republica Coreea
Amos C. Lee și Sunghoon Kwon
Departamentul de Inginerie Electronică, Universitatea Kyung Hee, Deongyeong-daero, Giheung-gu, Yongin-si, Gyeonggi-do, 17104, Republica Coreea
Suk-Heung Song, Seojoo Kim, Hyeli Kim și Wook Park
Institute of Entrepreneurial Bio Convergence, Seoul National University, 1, Gwanak-ro, Gwanak-gu, Seoul, 08826, Republica Coreea
Institutul Național de Cercetări Biomedice din Spitalul Universitar Național din Seul, Spitalul Universitar Național din Seul, 101, Daehak-ro Jongno-gu, Seul, 03080, Republica Coreea
Adresa curentă: Celemics Inc., 131, Gasandigital 1-ro, Geumcheon-gu, Seul, 08506, Republica Coreea
Taehoon Ryu și Parcul Jaejun
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Puteți căuta acest autor și în PubMed Google Scholar
Contribuții
Y.C., T.R., W.P. și S.K. a inițiat și conceput experimentele. Y.C., A.C.L., W.P. și S.K. a scris manuscrisul. Y.C., T.R., A.C.L., H.C., H.L., J.P., S.S., S.K. și H.K. a efectuat cercetarea, inclusiv sinteza și analiza ADN-ului.
Autori corespondenți
Declarații de etică
Interese concurente
Y.C., T.R., S.S., S.K., H.K., W.P. și S.K. sunt inventatori ai unei cereri de brevet pentru metoda descrisă în această lucrare. Ceilalți autori nu declară niciun conflict de interese.
Informatii suplimentare
Nota editorului: Springer Nature rămâne neutru în ceea ce privește revendicările jurisdicționale din hărțile publicate și afilierile instituționale.
- Laborator folosind un tranzistor pentru a controla încărcări de curent ridicat cu un Arduino; Calcul fizic ITP
- Impactul selecției mediilor și antispumante asupra producției și calității anticorpilor monoclonali utilizând un nivel ridicat
- Whiting Fish este bogat în Omega 3 sănătos
- Hoverboard Scooter Există beneficii pentru sănătate ale utilizării Hoverboard-urilor; King Power Boards - Canada; s
- Este legat de restaurantul dvs. să posteze meniul de informații nutriționale Trinfo